Οι υπολογιστικές υπηρεσίες του ΚΗΔ ΑΠΘ χρησιμοποιήθηκαν από το εργαστήριο του κ. Θ. Ι. Αμπατζόπουλου (Τμήμα Βιολογίας, Τομέας Γενετικής, Ανάπτυξης και Μοριακής Βιολογίας), στα πλαίσια της διδακτορικής διατριβής της Α. Λάγκη, με τίτλο «Χαρακτηρισμός και ταυτοποίηση καραβίδων εσωτερικών νερών Β. Ελλάδας». Μέρος των αποτελεσμάτων της εν λόγω διατριβής έχουν δημοσιευθεί στο περιοδικό “BMC Evolutionary Biology” με τίτλο “Microevolution of the noble crayfish (Astacus astacus) in the Southern Balkan Peninsula” (Laggis et al. 2017).
Στόχος της διδακτορικής διατριβής είναι η γενετική ταυτοποίηση, χαρακτηρισμός και δομή των καραβίδων που απαντώνται στον Ελλαδικό χώρο (Astacus astacus, A. leptodactylus, Austropotamobius torrentium και Pacifastacus leniusculus). Παράλληλα, έγινε περαιτέρω διερεύνηση των φυλογενετικών σχέσεων των καραβίδων στην Ευρώπη (γένη Astacus και Austropotamobius). Η προαναφερθείσα εργασία (Laggis et al. 2017), εστιάζει το ενδιαφέρον της τόσο στη γενετική δομή του Astacus astacus στην Ελλάδα, όσο και στη φυλογεωγραφία του είδους στην Ευρώπη. Για την διεκπεραίωση των παραπάνω στόχων και λόγο του όγκου των αναλύσεων, έγινε χρήση των υπηρεσιών του ΚΗΔ ΑΠΘ. Συγκεκριμένα, οι αναλύσεις που πραγματοποιήθηκαν στην Ιδρυματική συστοιχία του ΚΗΔ έκαναν χρήση των λογισμικών MsVar v. 1.3 (Storz & Beaumont 2002) και Beast v. 1.8 (Drummond et al. 2012). Περαιτέρω, χρησιμοποιήθηκαν οι γνώσεις και η τεχνική υποστήριξη του ανθρώπινου δυναμικού του ΚΗΔ ΑΠΘ, όσον αφορά την υπολογιστική υποδομή και τις γλώσσες προγραμματισμού (R και Python).
Τα αποτελέσματα της δημογραφικής ιστορίας του Astacus astacus στην Ελλάδα (Εικόνα 1), λήφθηκαν από την εκτέλεση των υπολογιστικών εργασιών στην Ιδρυματική συστοιχία του ΚΗΔ ΑΠΘ κάνοντας χρήση του λογισμικού MsVar. Οι υπολογιστικές εργασίες διήρκησαν τρείς (3) μήνες και ο συνολικός υπολογιστικός χρόνος (CPU time) ήταν 91.561 ώρες. Με άλλα λόγια, η ολοκλήρωση των απαιτούμενων υπολογισμών σε έναν μέσο επεξεργαστή θα απαιτούσε περίπου δέκα (10) έτη. Αντίστοιχα, τα φυλογενετικά δένδρα που προέκυψαν από το Beast (Εικόνα 2), έτρεξαν στις υποδομές του ΚΗΔ ΑΠΘ για περίπου έναν (1) μήνα, καταναλώνοντας συνολικά 11.569 CPU ώρες. Το τελευταίο, αντιστοιχεί περίπου σε χρονικό διάστημα 482 ημερών.
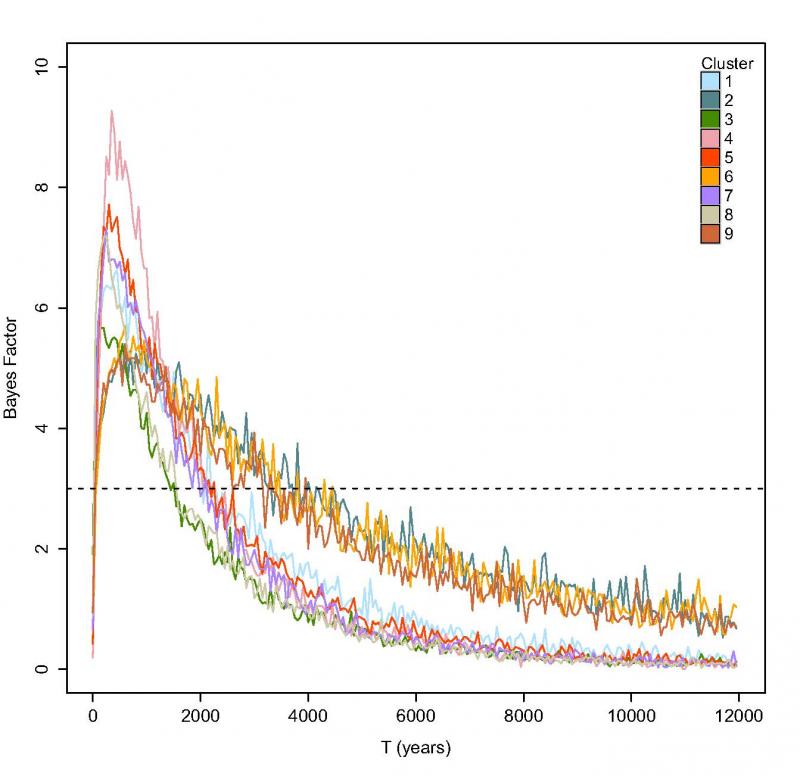
Εικόνα 1: Παράδειγμα αποτελέσματος της χρήσης του MsVar στην Ιδρυματική συστοιχία του ΚΗΔ ΑΠΘ (όπως δίνεται από Laggis et al., 2017).
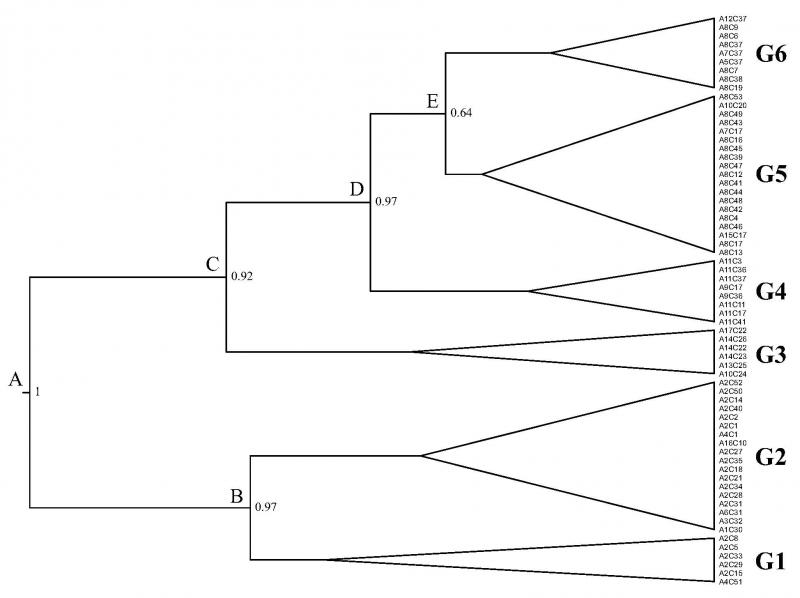
Εικόνα 2: Παράδειγμα φυλογενετικού δένδρου, που προέκυψε από τη χρήση του λογισμικού Beast στην Ιδρυματική συστοιχία του ΚΗΔ ΑΠΘ (όπως δίνεται από Laggis et al., 2017).
Βιβλιογραφία:
Laggis, A. et al., 2017. Microevolution of the noble crayfish (Astacus astacus) in the Southern Balkan Peninsula. BMC Evolutionary Biology, 17(1), p.122.
Storz, J.F. & Beaumont, M.A., 2002. Testing for genetic evidence of population expansion and contraction: an empirical analysis of microsatellite DNA variation using a hierarchical Bayesian model. Evolution, 56(1), pp.154–166.
Drummond, A.J. et al., 2012. Bayesian phylogenetics with BEAUti and the BEAST 1.7. Molecular Biology and Evolution, 29(8), pp.1969–1973.

